Spotlight - AG Oehme
Erstellung von Medikamentenempfindlichkeitsprofilen in INFORM
Das internationale multizentrische Programm INdividualized therapy FOr high-Risk childhood Malignancies (INFORM) für pädiatrische Präzisionsonkologie befasst sich mit einem ungedeckten medizinischen Bedarf durch die Bereitstellung umfassender molekularer Next-Generation-Sequenzierungsinformationen (NGS), einschließlich flächendeckender Ganzgenomsequenzierung, Ganz-Exom-Sequenzierung, RNA-Sequenzierung und 850k-DNA-Methylierungsprofilierung für rezidivierte/refraktäre pädiatrische Krebspatienten.
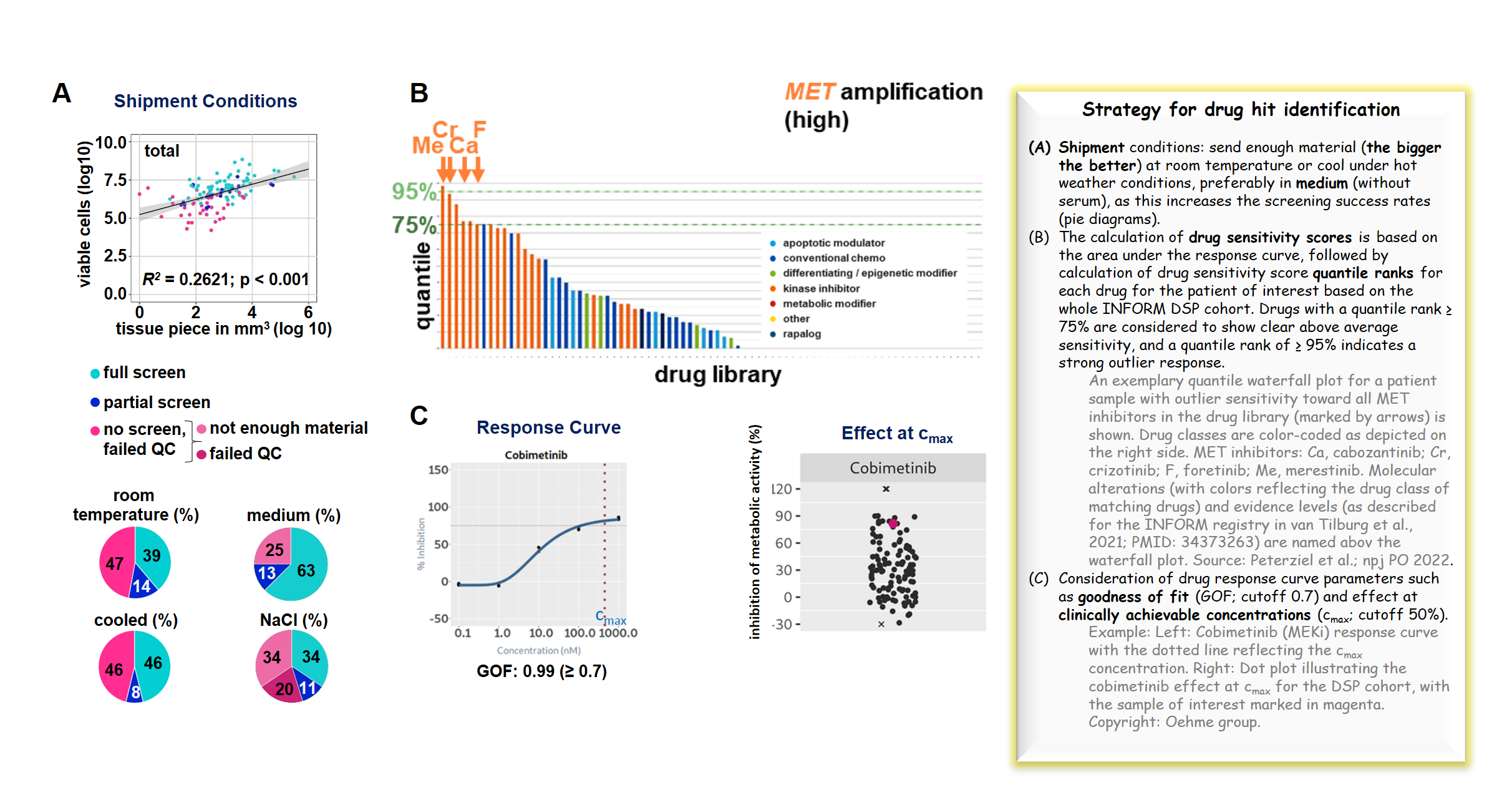
Um INFORM eine weitere funktionelle Komponente hinzuzufügen, haben wir eine personalisierte Plattform zur Erstellung von Profilen der Medikamentensensitivität (DSP) auf der Grundlage von Messungen der Stoffwechselaktivität eingeführt, die die NGS-basierte Zielidentifizierung ergänzen. Wir haben kürzlich die Ergebnisse einer zweijährigen Pilotstudie veröffentlicht, in der wir ex vivo DSP an frischen Tumorproben durchgeführt haben, die wir mit INFORM gewonnen haben (Peterziel et al. 2022, NPJ Precision Oncology, PMID 36575299). Wir erstellten Protokolle für die diagnosespezifische Probenbehandlung, das Wirkstoffscreening mit einer Bibliothek klinisch relevanter Wirkstoffe und die DSP-Analyse. Das Wirkstoffscreening wurde an von Patienten stammenden multizellulären 3D-Frischgewebekulturen innerhalb kurzer Zeit (in den meisten Fällen zwei bis sieben Tage) nach Annahme der Proben durchgeführt. Bei einigen Zufallsfällen bestätigten wir, dass diese Kulturen noch weitgehend die multizelluläre Zusammensetzung des ursprünglichen Tumors widerspiegelten. Das eingesandte Material war in der Mehrzahl der Fälle quantitativ und qualitativ ausreichend, um DSP-Befunde mit einer Durchlaufzeit von etwa drei Wochen zu melden. Die DSP-Ergebnisse stimmten mit den identifizierten molekularen Zielen überein, einschließlich BRAF-, ALK-, MET- und TP53-Status. Dies wurde besonders deutlich, als die Erkennung von Ausreißern (innerhalb der DSP-Kohorte) in den Ansatz zur Auswahl von Medikamentenhits einbezogen wurde. Als Mehrwert der molekularen Daten wurden darüber hinaus in der Mehrzahl der Fälle, in denen keine (sehr) aussagekräftigen molekularen Ereignisse vorlagen, Schwachstellen in den Medikamenten identifiziert. Mit dem Ziel, erweiterte Ansätze für die Arzneimittelprüfung und gegebenenfalls die Entwicklung klinischer Studien voranzutreiben, etablieren wir organoidähnliche Langzeit-Zellkulturen und, in Zusammenarbeit mit anderen KiTZ-Gruppen, murine patient-derived xenograft (mPDX)-Modelle der eingereichten INFORM-Frischgewebeproben.
Insgesamt hat unsere Proof-of-Concept-Pilotstudie bewiesen, dass DSP innerhalb eines klinisch relevanten Zeitraums durchführbar ist und wertvolle funktionelle Informationen liefert, wenn sie in Präzisions-Onkologie-Programme wie INFORM einbezogen wird.
Zebrafisch-Xenograft-Modelle für das Wirkstoffscreening
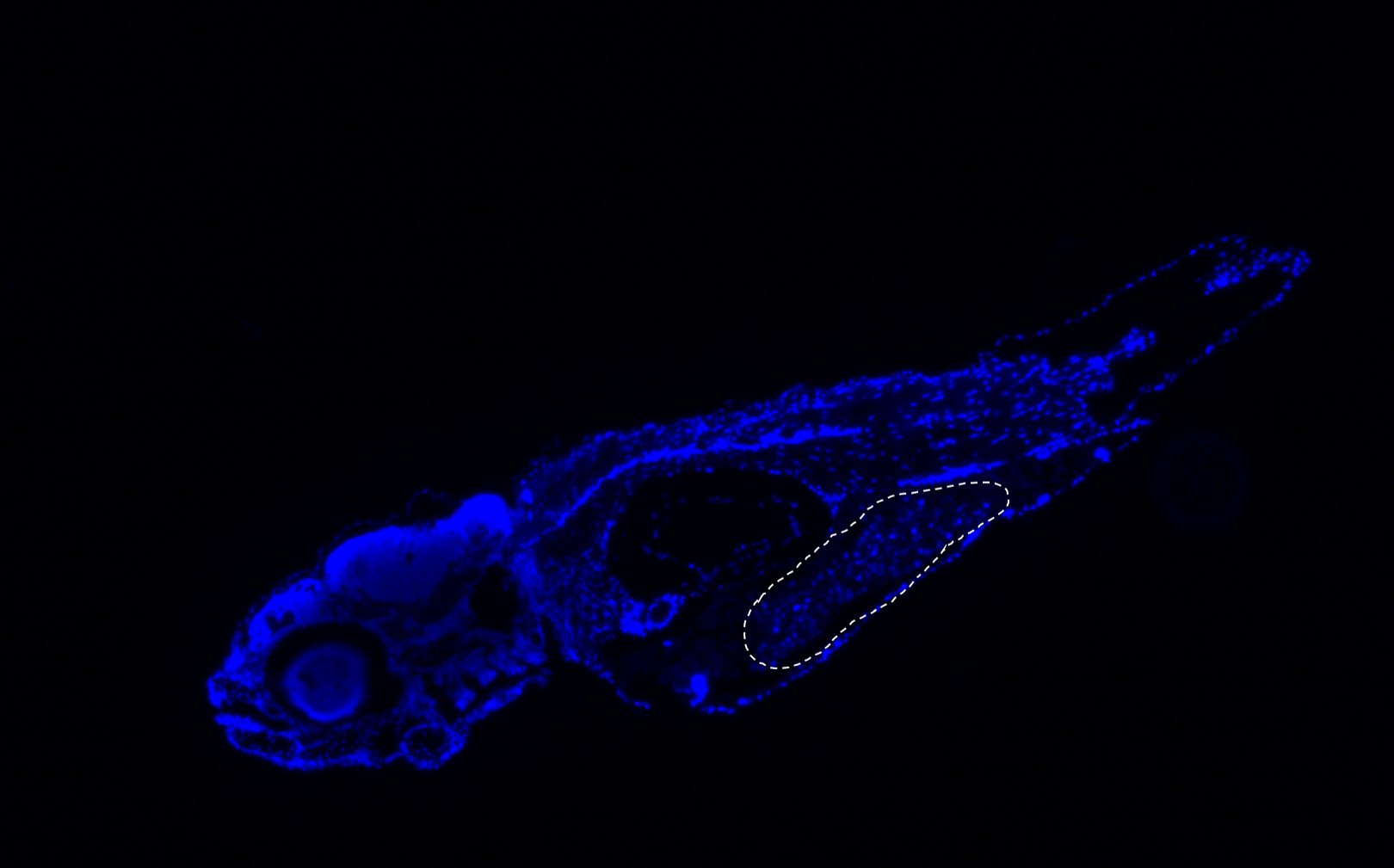
Wir haben ein Zebrafisch-Embryo (Danio rerio) als Xenograft-Modell für pädiatrische Tumorzellen etabliert, um das Tumorwachstum und die Tumorprogression (z. B. Migration) zu untersuchen. Dieses System basiert auf der Mikroinjektion von pädiatrischen (von Patienten stammenden) Tumorzellen in den Dottersack und den perivitellinen Raum (PVS). Darüber hinaus etablieren wir zusätzlich ein orthotopes pädiatrisches Hirntumor-Zebrafisch-Modell durch Mikroinjektion von Zellen in Zebrafisch-Blastulae.
Funktionelle Analyse der Therapieresistenz
Obwohl die Überlebenschancen für Kinder mit bösartigen Krebserkrankungen in den letzten Jahrzehnten erheblich gestiegen sind, können immer noch zu viele Kinder nicht geheilt werden und erliegen ihrer Krankheit. In vielen Fällen ist dies entweder darauf zurückzuführen, dass es bei bestimmten Tumorentitäten keine geeigneten Behandlungsmöglichkeiten gibt oder dass eine Therapieresistenz entweder vor der Behandlung besteht oder sich während der Therapie entwickelt. Das Ziel unserer Projekte ist die Identifizierung handlungsfähiger zellulärer Veränderungen in pädiatrischen Tumoren des Nervensystems (Neuroblastom, Medulloblastom, Gliom, Ependymom) durch Hochdurchsatz-Wirkstoffscreening.
Bei behandlungsresistenten Neuroblastomen untersuchen wir, wie die Hemmung von Zelloberflächen-Wachstumsrezeptoren, Medikamenten-Efflux-Pumpen und extrazellulären Matrixkomponenten (z. B. MMPs) resistente Zellen für eine Chemotherapie sensibilisieren kann. Ein weiterer Schwerpunkt unserer Studien ist das Lysosom, eine zelluläre Organelle, die für den Abbau von zellulären Makromolekülen verantwortlich ist. Eine Reihe von Studien legt nahe, dass Lysosomen durch verschiedene Mechanismen zur Chemoresistenz von Krebszellen beitragen, darunter Autophagie und lysosomale Exozytose. Durch die systematische Korrelation von Hochdurchsatz-Wirkstoffscreens mit Daten aus der Hochkontrastmikroskopie untersuchen wir, wie häufig lysosomale Resistenzmechanismen bei Tumoren des pädiatrischen Nervensystems auftreten und ob sie als Ziele für Kombinationstherapien genutzt werden können.
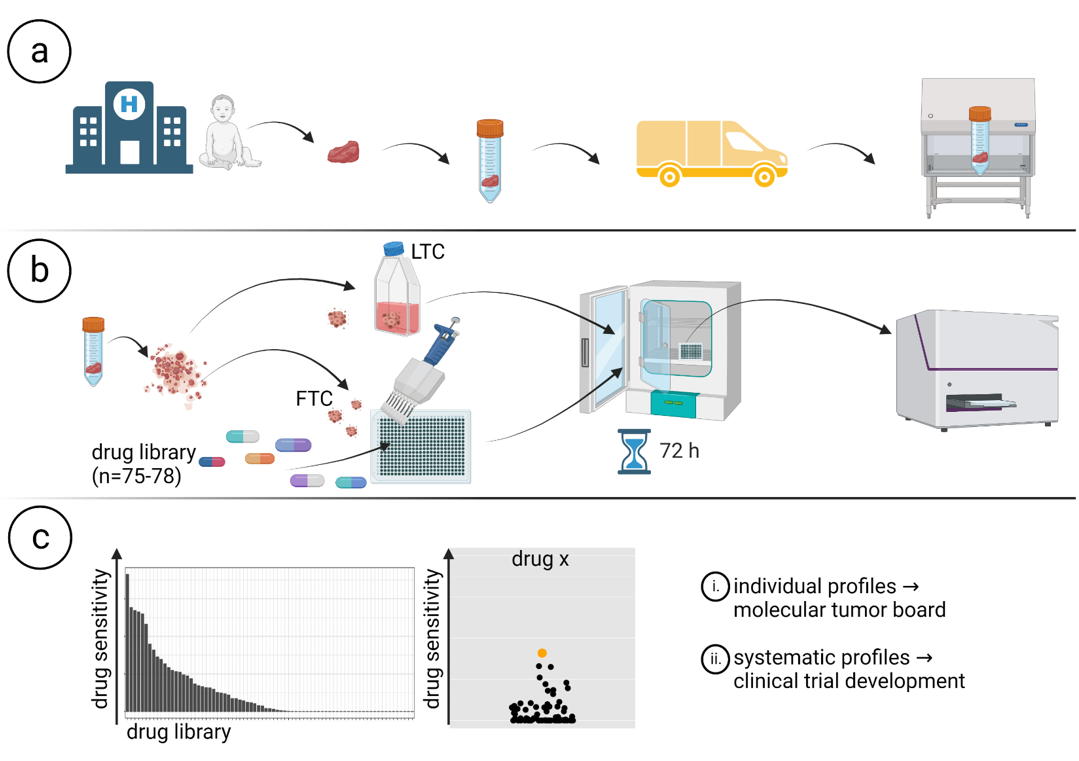
Arbeitsablauf der personalisierten Arzneimittelempfindlichkeitsprofilierung von INFORM. Erstellt mit BioRender.com. (a) Probenentnahme und -versand. (b) Erzeugung von patienteneigenen ex vivo Frischgewebe-Sphäroid-Kulturen (FTCs) und Behandlung mit einer Medikamentenbibliothek. Ablesung: Stoffwechselaktivität. (c) Datenerfassung, Analyse und Erstellung von Berichten über die Empfindlichkeit von Medikamenten. Quelle: Peterziel et al.; npj PO 2022.
- Celikyürekli, Simay (Ph.D. student)
- Digel, Lea (student assistant)
- Friedenauer, Aileen (technical assistant)
- Gatzweiler, Charlotte (MD student)
- Gerloff, Xenia (student assistant)
- Herter, Sonja (Ph.D. student)
- Hugo, Anette (technical assistant)
- Körholz, Katharina (MD student)
- Loboda, Anna (Ph.D. student)
- Melero Emperador, Marta (Ph.D. student)
- Mohr, Jacqueline (student assistant)
- Müller, Michael (MD student)
- Najafi, Sara Dr. sc. hum. (Post-Doc)
- Oehme, Ina PD Dr. phil. nat. (group leader)
- Peterziel, Heike Dr. rer. nat. (staff scientist)
- Rösch, Lisa (Ph.D. student)
- Salem-Altintas, Rabia (MD student)
- Seiboldt, Till (MD student)
- Stroh-Dege, Alex (technical assistant)
- Weidmann, Marko (technical assistant)
- Zeiser, Constantia (MD student)