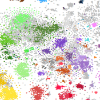
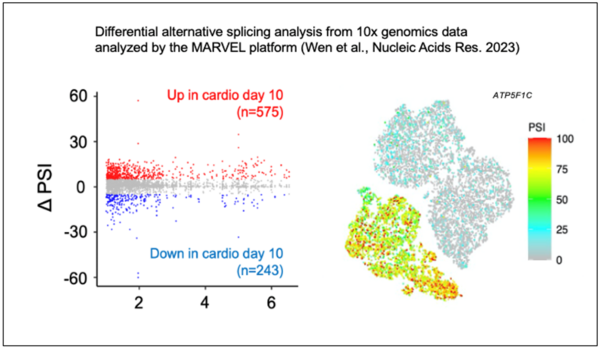
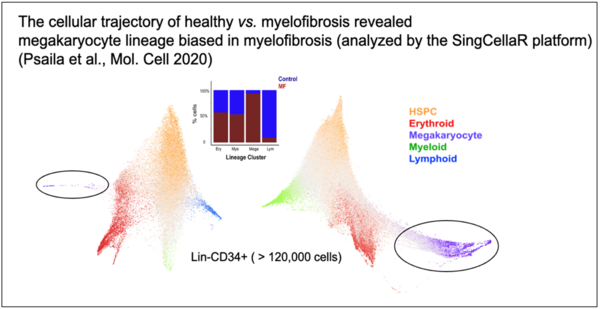
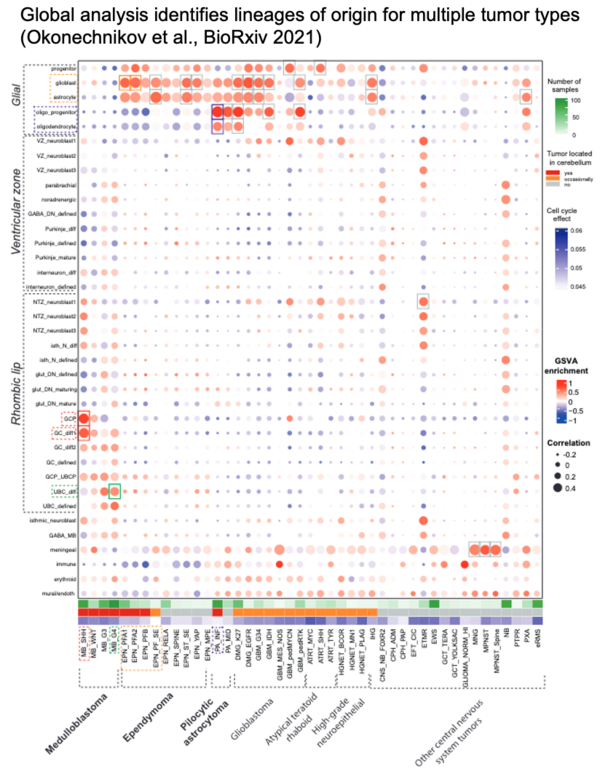
Wir haben robuste Analysemethoden und Plattformen entwickelt, um die scRNA-Seq zu erleichtern. Zu diesen Plattformen gehört SingCellaR (Roy et al., Cell Rep. 2021; Wang et al., STAR Protoc. 2022), ein umfassendes Analysetool zur Integration, Abfrage und Visualisierung von scRNA-seq. VALERIE (Wen et al., PLoS Comput. Biol. 2020) unterstützt die Visualisierung von alternativen Spleißereignissen bei Einzelzellauflösung. MARVEL (Wen et al., Nucleic Acids Res. 2023) unterstützt die Einzelzell-Spleißanalyse von scRNA-seq, die mit platten- und tröpfchenbasierten Methoden erzeugt wurden. MARVEL ermöglicht eine systematische und integrierte Analyse von Spleißen und Genexpression, um die Spleißlandschaft zu charakterisieren.
Wir haben mit experimentellen Mitarbeitern eine neuartige Methode zur Integration von scRNA-seq und hochempfindlicher Mutationsdetektion aus derselben Einzelzelle entwickelt, um Krebsstammzellen bei chronisch-myeloischer Leukämie zu untersuchen (Giustacchini & Thongjuea et al., Nat. Med. 2017). Wir verbesserten die Hochdurchsatzkapazität zum Nachweis mehrerer gezielter Mutationen (TARGET-seq) (Rodriguez-Meira et al., Mol Cell. 2019). Wir analysierten 10x-Genomikdaten, um dynamische Veränderungen in der zellulären Zusammensetzung aufzuzeigen, und identifizierten orts- und entwicklungsstadienspezifische Transkriptions- und Genregulationsnetzwerke über menschliche Entwicklungsstadien hinweg (Roy et al., Cell Rep. 2021). Wir haben an großen Einzelzelldaten von Myelofibrose-Patienten gearbeitet und die starke Abstammungsorientierung in Richtung Megakaryozyten-Differenzierung im Stammzell-/Progenitor-Kompartiment aufgezeigt, was zur Entdeckung von G6B als potenziellem Immuntherapieziel führte (Psaila et al., Mol Cell. 2020).
Das Team arbeitet eng mit den PIs des KiTZ an mehreren Projekten zur Einzelzell-/Raumanalyse von Medulloblastomen (Okonechnikov et al., BioRxiv 2021; Ghasemi et al., BioRxiv 2022; Okonechnikov et al., Acta Neuropathol. Commun. 2023) und hochgradige Gliome. Wir leiten das Teilprojekt 1 des HEROES-AYA-Projekts, das sich mit der Anwendung modernster Einzelzelltechnologien zur Charakterisierung der Sarkom-Heterogenität anhand von Einzelzellgenomen und Epigenomen befasst. Wir arbeiten auch mit Teilprojekt 3 zusammen, das sich mit nicht-invasiven Technologien zur Erkennung und Überwachung von Sarkomen befasst. Wir haben uns mehr auf die Integration von Einzelzell- und räumlichen Ansätzen für die Analyse der Zell-Zell-Kommunikation in verschiedenen Zelltypen bei Hirntumoren konzentriert und konzentrieren uns auch auf die Analyse des alternativen Spleißens bei verschiedenen Krebsarten.